生存分析(2)_代码实例
前言
最近在学习生存分析,为了防止自己忘记常见的生存分析步骤以及 survival 包中的一些函数用法,故于此记录。
本篇文章以 survival 包中的内置数据集为例,介绍生存分析的一般步骤,如有错误请在评论区指出。
1. 平台介绍
survival包: v3.2.13
R version: 4.1.2 (2021-11-01)
IDE: RStudio 2022.02.3
2. 数据处理
可以使用命令 data(package = 'survival') 来查询 survival 包中的内置数据集,本例中以膀胱癌数据集 bladder 为例, 输入 ?bladder 查看内置数据集的详细信息。bladder数据集内容如下图所示:

| 变量名 | 解释 |
|---|---|
| id | 病人编号 |
| rx | 治疗方式,1为安慰剂,2为三胺硫磷 |
| number | 初始肿瘤数目(8 = 8及8以上) |
| size | 最大初始肿瘤的大小(厘米) |
| stop | 观察持续时间(开始为0) |
| event | 是否治愈,1为治愈,0为未治愈 |
| enum | 不知道是什么意思 |
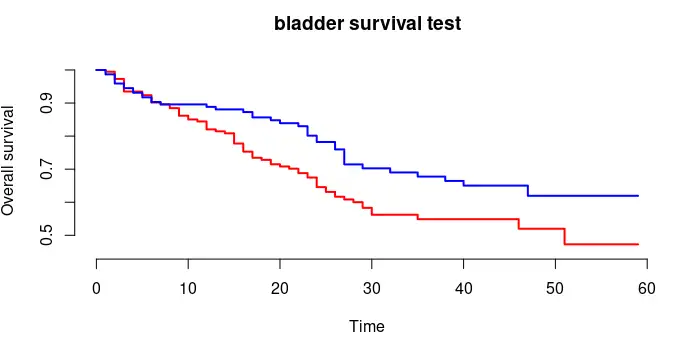
我们的目标是按照rx变量分组画出两条生存曲线,并对这两条生存曲线进行统计学检验,检查两种治疗方式是否存在有统计学上的差异。即最后的目标图如下:

3. 绘制生存曲线
代码如下
library(survival)
# bladder是内置数据集,将其赋给bladder变量
bladder <- bladder
# 构建一个生存对象,stop是时间,event是生死状态
surv_data <- survfit(
Surv(stop, event) ~ rx,
data = bladder
)
# 开始绘图
plot(
surv_data,
col = c('red', 'blue'),
xlab = 'Time',
ylab = 'Overall survival',
main = 'bladder survival test'
)
结果如图所示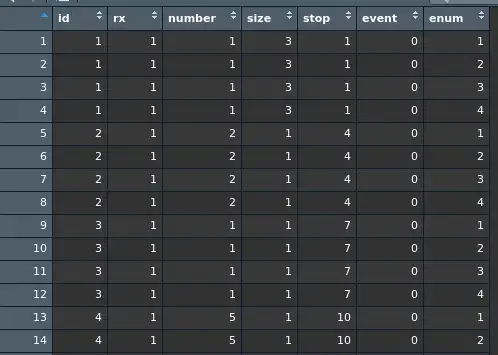
然后对这两条生存曲线进行统计学检验,生存曲线常用的检验方法为 Logrank 检验 。可以用 survdiff 函数进行检验,代码如下
temp <- survdiff(
formula = Surv(stop, event) ~ rx,
data = bladder
)
# 用卡方分布,自由度手动计算p值
p_value <- 1 - pchisq(temp$chisq, length(temp$n) - 1)
再对图片进行一个美化
plot(
surv_data,
col = c('red', 'blue'),
xlim = c(0, 60), # 设置x变量显示范围
ylim = c(0.4, 1.0), # 设置y变量显示范围
lwd = 2, # 曲线粗细
xaxt = 'n', # 关闭x轴刻度
yaxt = 'n', # 关闭y轴刻度
bty = 'n', # 关闭外框线
xlab = 'Time',
ylab = 'Overall survival',
main = 'bladder survival test'
)
## 绘制坐标轴
axis(
side = 1, # 横坐标
seq(from = 0, to = 60, by = 10) # 设置刻度间隔
)
axis(
side = 2, # 纵坐标
seq(from = 0.4, to = 1.0, by = 0.2) # 设置刻度间隔
)
## 添加图例
legend(
x = 50,
y = 0.95,
legend = c('药', '安慰剂'),
col = c('blue', 'red'),
lwd = 3, # 设置标签的线
bty = 'n', # 去除标签的外框线
)
## 添加检验结果
legend(
x = 0,
y = 0.5,
legend = paste('Log rank p=', round(p_value, 3), sep = ''),
bty = 'n',
cex = 0.9
)
结果如下图